- Press and hold down the left mouse button and move the mouse to rotate.
- Press and hold down the central mouse button and move the mouse to translate.
- Use the mouse wheel to zoom in or out.
- Click on any atom on your molecule and the corresponding residue label will be placed beneath the viewer.
| Color | ConSurf Grade |
| No Conservation data available | |
| 1 | |
| 2 | |
| 3 | |
| 4 | |
| 5 | |
| 6 | |
| 7 | |
| 8 | |
| 9 |
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
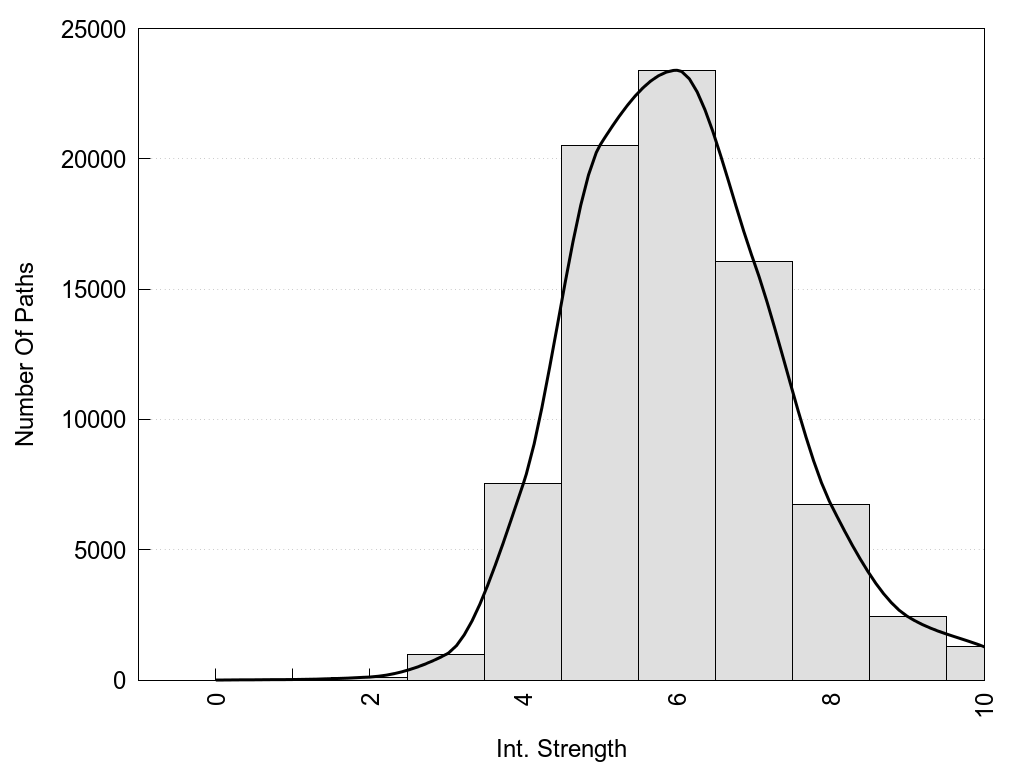
Int. Strength: the interaction strength between the two nodes.
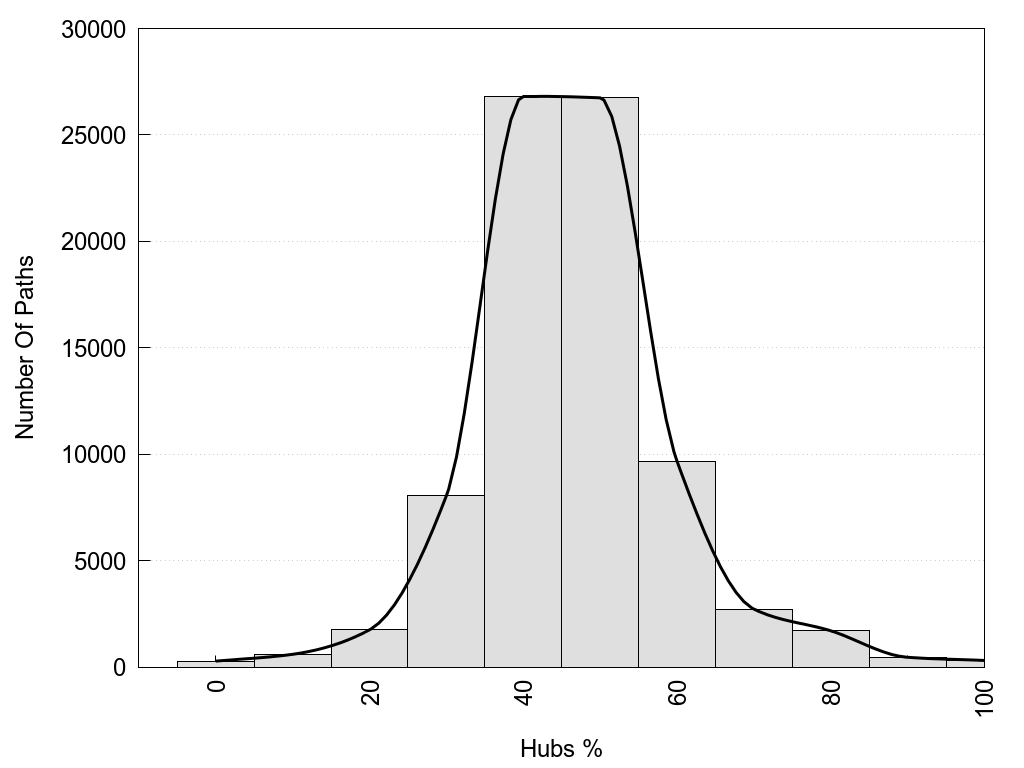
Hub1?, Hub2?: "Yes" if the corresponding node has more than 3 links, otherwise "No".
Community: the id of the community the link belong to, otherwise 0.
ConSurf1, ConSurf2: these columns report the ConSurf conservation grades of the two nodes involved in a link.
| Index | Node1 | Node2 | Int. Strength | Hub1? | Hub2? | Community | ConSurf1 | ConSurf2 |
|---|---|---|---|---|---|---|---|---|
| 1 | L:L:P2 | L:L:Y1 | 9.74 | Yes | Yes | 1 | 0 | 0 |
| 2 | L:L:I31 | L:L:Y1 | 3.63 | No | Yes | 1 | 0 | 0 |
| 3 | L:L:Y1 | R:R:F199 | 6.19 | Yes | Yes | 1 | 0 | 3 |
| 4 | L:L:Y1 | R:R:D200 | 8.05 | Yes | Yes | 1 | 0 | 4 |
| 5 | L:L:Y1 | R:R:R208 | 12.35 | Yes | Yes | 1 | 0 | 4 |
| 6 | L:L:L30 | L:L:P2 | 4.93 | No | Yes | 1 | 0 | 0 |
| 7 | L:L:P2 | R:R:F286 | 7.22 | Yes | No | 1 | 0 | 4 |
| 8 | L:L:P2 | R:R:I292 | 3.39 | Yes | No | 0 | 0 | 4 |
| 9 | L:L:K4 | L:L:P5 | 3.35 | No | No | 0 | 0 | 0 |
| 10 | L:L:K4 | L:L:Y27 | 8.36 | No | No | 0 | 0 | 0 |
| 11 | L:L:K4 | R:R:N186 | 8.39 | No | No | 0 | 0 | 1 |
| 12 | L:L:N7 | L:L:Y20 | 3.49 | No | Yes | 3 | 0 | 0 |
| 13 | L:L:N7 | R:R:V187 | 4.43 | No | No | 3 | 0 | 2 |
| 14 | L:L:N7 | R:R:T188 | 16.08 | No | No | 3 | 0 | 3 |
| 15 | L:L:D16 | L:L:P8 | 8.05 | No | No | 0 | 0 | 0 |
| 16 | L:L:P8 | L:L:Y20 | 8.34 | No | Yes | 0 | 0 | 0 |
| 17 | L:L:D11 | L:L:E10 | 10.39 | No | No | 0 | 0 | 0 |
| 18 | L:L:D16 | L:L:P13 | 11.27 | No | No | 0 | 0 | 0 |
| 19 | L:L:M17 | L:L:Y21 | 16.76 | No | Yes | 3 | 0 | 0 |
| 20 | L:L:M17 | R:R:F28 | 3.73 | No | Yes | 3 | 0 | 6 |
| 21 | L:L:Y20 | L:L:Y21 | 4.96 | Yes | Yes | 3 | 0 | 0 |
| 22 | L:L:Y20 | R:R:T188 | 7.49 | Yes | No | 3 | 0 | 3 |
| 23 | L:L:Y21 | R:R:F28 | 9.28 | Yes | Yes | 3 | 0 | 6 |
| 24 | L:L:Y21 | R:R:E29 | 5.61 | Yes | Yes | 0 | 0 | 4 |
| 25 | L:L:Y21 | R:R:D190 | 9.2 | Yes | No | 3 | 0 | 1 |
| 26 | L:L:I28 | L:L:L24 | 5.71 | No | No | 0 | 0 | 0 |
| 27 | L:L:L24 | R:R:E29 | 9.28 | No | Yes | 0 | 0 | 4 |
| 28 | L:L:R25 | R:R:E29 | 16.28 | No | Yes | 0 | 0 | 4 |
| 29 | L:L:R25 | R:R:D32 | 9.53 | No | No | 0 | 0 | 8 |
| 30 | L:L:H26 | R:R:T295 | 10.95 | No | No | 0 | 0 | 1 |
| 31 | L:L:H26 | R:R:C296 | 8.85 | No | No | 0 | 0 | 2 |
| 32 | L:L:I31 | L:L:Y27 | 12.09 | No | No | 0 | 0 | 0 |
| 33 | L:L:Y27 | R:R:P183 | 4.17 | No | Yes | 0 | 0 | 1 |
| 34 | L:L:I28 | R:R:D104 | 7 | No | No | 0 | 0 | 4 |
| 35 | L:L:I28 | R:R:F184 | 8.79 | No | No | 0 | 0 | 1 |
| 36 | L:L:N29 | R:R:D104 | 4.04 | No | No | 0 | 0 | 4 |
| 37 | L:L:N29 | R:R:N299 | 9.54 | No | No | 0 | 0 | 4 |
| 38 | L:L:L30 | R:R:F286 | 7.31 | No | No | 1 | 0 | 4 |
| 39 | L:L:L30 | R:R:H298 | 9 | No | No | 0 | 0 | 3 |
| 40 | L:L:I31 | R:R:F199 | 5.02 | No | Yes | 1 | 0 | 3 |
| 41 | L:L:T32 | R:R:Y100 | 8.74 | No | No | 0 | 0 | 6 |
| 42 | L:L:R33 | R:R:F282 | 7.48 | Yes | Yes | 1 | 0 | 5 |
| 43 | L:L:R33 | R:R:N283 | 4.82 | Yes | Yes | 1 | 0 | 5 |
| 44 | L:L:R33 | R:R:F286 | 8.55 | Yes | No | 1 | 0 | 4 |
| 45 | L:L:R33 | R:R:F302 | 19.24 | Yes | No | 1 | 0 | 5 |
| 46 | L:L:Q34 | R:R:T97 | 9.92 | Yes | Yes | 1 | 0 | 6 |
| 47 | L:L:Q34 | R:R:Y100 | 13.53 | Yes | No | 1 | 0 | 6 |
| 48 | L:L:Q34 | R:R:W106 | 3.29 | Yes | Yes | 1 | 0 | 8 |
| 49 | L:L:Q34 | R:R:N116 | 3.96 | Yes | Yes | 1 | 0 | 5 |
| 50 | L:L:Q34 | R:R:Q120 | 6.4 | Yes | Yes | 1 | 0 | 6 |
| 51 | L:L:?36 | L:L:R35 | 21.56 | Yes | Yes | 1 | 0 | 0 |
| 52 | L:L:R35 | R:R:F173 | 5.34 | Yes | Yes | 0 | 0 | 5 |
| 53 | L:L:R35 | R:R:T212 | 9.06 | Yes | No | 1 | 0 | 5 |
| 54 | L:L:R35 | R:R:N283 | 12.05 | Yes | Yes | 1 | 0 | 5 |
| 55 | L:L:R35 | R:R:D287 | 8.34 | Yes | No | 1 | 0 | 4 |
| 56 | L:L:?36 | R:R:T97 | 6.23 | Yes | Yes | 1 | 0 | 6 |
| 57 | L:L:?36 | R:R:Q120 | 6.75 | Yes | Yes | 1 | 0 | 6 |
| 58 | L:L:?36 | R:R:I124 | 6.03 | Yes | Yes | 1 | 0 | 7 |
| 59 | L:L:?36 | R:R:L215 | 4.68 | Yes | No | 1 | 0 | 5 |
| 60 | L:L:?36 | R:R:Q219 | 15.75 | Yes | No | 1 | 0 | 6 |
| 61 | L:L:?36 | R:R:N283 | 3.48 | Yes | Yes | 1 | 0 | 5 |
| 62 | L:L:?36 | R:R:H306 | 7.61 | Yes | Yes | 1 | 0 | 6 |
| 63 | R:R:D190 | R:R:F28 | 7.17 | No | Yes | 3 | 1 | 6 |
| 64 | R:R:E29 | R:R:Y192 | 8.98 | Yes | No | 0 | 4 | 1 |
| 65 | R:R:D31 | R:R:N30 | 9.42 | No | No | 0 | 5 | 6 |
| 66 | R:R:D32 | R:R:L35 | 10.86 | No | No | 0 | 8 | 3 |
| 67 | R:R:C296 | R:R:C33 | 7.28 | No | No | 0 | 2 | 8 |
| 68 | R:R:L35 | R:R:P36 | 4.93 | No | No | 0 | 3 | 4 |
| 69 | R:R:M39 | R:R:P36 | 6.71 | No | No | 0 | 4 | 4 |
| 70 | R:R:I40 | R:R:P36 | 6.77 | No | No | 0 | 3 | 4 |
| 71 | R:R:F41 | R:R:L45 | 9.74 | No | No | 0 | 6 | 5 |
| 72 | R:R:L94 | R:R:Y47 | 7.03 | Yes | Yes | 1 | 7 | 7 |
| 73 | R:R:F98 | R:R:Y47 | 9.28 | No | Yes | 0 | 6 | 7 |
| 74 | R:R:L303 | R:R:Y47 | 4.69 | No | Yes | 0 | 4 | 7 |
| 75 | R:R:H306 | R:R:Y47 | 4.36 | Yes | Yes | 1 | 6 | 7 |
| 76 | R:R:L307 | R:R:Y47 | 5.86 | No | Yes | 1 | 5 | 7 |
| 77 | R:R:M310 | R:R:Y47 | 7.18 | Yes | Yes | 1 | 7 | 7 |
| 78 | R:R:M310 | R:R:V50 | 15.22 | Yes | No | 0 | 7 | 7 |
| 79 | R:R:I51 | R:R:L94 | 8.56 | No | Yes | 1 | 7 | 7 |
| 80 | R:R:I51 | R:R:P95 | 3.39 | No | No | 1 | 7 | 8 |
| 81 | R:R:G54 | R:R:L53 | 3.42 | No | No | 0 | 7 | 5 |
| 82 | R:R:G57 | R:R:S56 | 3.71 | No | No | 0 | 8 | 5 |
| 83 | R:R:L61 | R:R:N58 | 4.12 | No | Yes | 4 | 9 | 9 |
| 84 | R:R:N58 | R:R:S83 | 5.96 | Yes | No | 4 | 9 | 9 |
| 85 | R:R:D86 | R:R:N58 | 18.85 | No | Yes | 0 | 9 | 9 |
| 86 | R:R:N58 | R:R:P317 | 14.66 | Yes | No | 0 | 9 | 9 |
| 87 | R:R:L59 | R:R:L87 | 4.15 | No | No | 0 | 4 | 8 |
| 88 | R:R:L61 | R:R:S83 | 6.01 | No | No | 4 | 9 | 9 |
| 89 | R:R:I62 | R:R:S83 | 6.19 | No | No | 0 | 7 | 9 |
| 90 | R:R:I65 | R:R:V80 | 4.61 | No | No | 0 | 7 | 5 |
| 91 | R:R:F327 | R:R:I65 | 6.28 | Yes | No | 0 | 8 | 7 |
| 92 | R:R:E70 | R:R:Q68 | 10.19 | No | No | 0 | 5 | 5 |
| 93 | R:R:D330 | R:R:Q68 | 5.22 | No | No | 0 | 5 | 5 |
| 94 | R:R:E70 | R:R:N76 | 6.57 | No | No | 0 | 5 | 8 |
| 95 | R:R:R72 | R:R:W148 | 5 | No | No | 0 | 6 | 5 |
| 96 | R:R:N73 | R:R:N76 | 14.98 | No | No | 0 | 8 | 8 |
| 97 | R:R:E137 | R:R:V74 | 5.7 | Yes | No | 0 | 8 | 6 |
| 98 | R:R:A155 | R:R:V74 | 3.39 | No | No | 0 | 7 | 6 |
| 99 | R:R:E137 | R:R:T75 | 7.06 | Yes | No | 0 | 8 | 7 |
| 100 | R:R:F327 | R:R:N76 | 4.83 | Yes | No | 0 | 8 | 8 |
| 101 | R:R:I77 | R:R:Y156 | 4.84 | No | No | 0 | 5 | 3 |
| 102 | R:R:L133 | R:R:L78 | 4.15 | No | Yes | 0 | 7 | 7 |
| 103 | R:R:I134 | R:R:L78 | 5.71 | No | Yes | 0 | 8 | 7 |
| 104 | R:R:E137 | R:R:L78 | 6.63 | Yes | Yes | 0 | 8 | 7 |
| 105 | R:R:I79 | R:R:Y320 | 3.63 | No | Yes | 0 | 8 | 9 |
| 106 | R:R:I159 | R:R:N81 | 12.74 | No | No | 0 | 7 | 9 |
| 107 | R:R:N81 | R:R:W163 | 6.78 | No | No | 0 | 9 | 9 |
| 108 | R:R:L82 | R:R:S130 | 7.51 | Yes | No | 0 | 9 | 8 |
| 109 | R:R:L131 | R:R:L82 | 5.54 | No | Yes | 2 | 8 | 9 |
| 110 | R:R:L82 | R:R:N316 | 15.1 | Yes | No | 2 | 9 | 9 |
| 111 | R:R:L82 | R:R:Y320 | 3.52 | Yes | Yes | 2 | 9 | 9 |
| 112 | R:R:S127 | R:R:S85 | 4.89 | No | No | 0 | 9 | 8 |
| 113 | R:R:D86 | R:R:T313 | 5.78 | No | No | 0 | 9 | 8 |
| 114 | R:R:D86 | R:R:N316 | 4.04 | No | No | 0 | 9 | 9 |
| 115 | R:R:C93 | R:R:V89 | 6.83 | No | Yes | 1 | 7 | 7 |
| 116 | R:R:S123 | R:R:V89 | 3.23 | No | Yes | 0 | 8 | 7 |
| 117 | R:R:I124 | R:R:V89 | 4.61 | Yes | Yes | 1 | 7 | 7 |
| 118 | R:R:T313 | R:R:V89 | 3.17 | No | Yes | 0 | 8 | 7 |
| 119 | R:R:F96 | R:R:M92 | 7.46 | Yes | Yes | 1 | 6 | 5 |
| 120 | R:R:M92 | R:R:N116 | 8.41 | Yes | Yes | 1 | 5 | 5 |
| 121 | R:R:M92 | R:R:V119 | 4.56 | Yes | No | 0 | 5 | 5 |
| 122 | R:R:M92 | R:R:Q120 | 4.08 | Yes | Yes | 1 | 5 | 6 |
| 123 | R:R:C93 | R:R:Q120 | 7.63 | No | Yes | 1 | 7 | 6 |
| 124 | R:R:C93 | R:R:I124 | 3.27 | No | Yes | 1 | 7 | 7 |
| 125 | R:R:L94 | R:R:P95 | 8.21 | Yes | No | 1 | 7 | 8 |
| 126 | R:R:H306 | R:R:L94 | 3.86 | Yes | Yes | 1 | 6 | 7 |
| 127 | R:R:L94 | R:R:M310 | 4.24 | Yes | Yes | 1 | 7 | 7 |
| 128 | R:R:F108 | R:R:F96 | 10.72 | Yes | Yes | 1 | 7 | 6 |
| 129 | R:R:F96 | R:R:M112 | 3.73 | Yes | No | 1 | 6 | 6 |
| 130 | R:R:F96 | R:R:N116 | 15.71 | Yes | Yes | 1 | 6 | 5 |
| 131 | R:R:Q120 | R:R:T97 | 4.25 | Yes | Yes | 1 | 6 | 6 |
| 132 | R:R:H306 | R:R:T97 | 5.48 | Yes | Yes | 1 | 6 | 6 |
| 133 | R:R:F98 | R:R:L102 | 4.87 | No | No | 0 | 6 | 4 |
| 134 | R:R:W106 | R:R:Y100 | 13.5 | Yes | No | 1 | 8 | 6 |
| 135 | R:R:H105 | R:R:M103 | 14.44 | No | No | 0 | 5 | 3 |
| 136 | R:R:M103 | R:R:V107 | 9.13 | No | No | 0 | 3 | 2 |
| 137 | R:R:H105 | R:R:K195 | 14.41 | No | No | 0 | 5 | 3 |
| 138 | R:R:F108 | R:R:W106 | 18.04 | Yes | Yes | 1 | 7 | 8 |
| 139 | R:R:C113 | R:R:W106 | 10.45 | No | Yes | 1 | 9 | 8 |
| 140 | R:R:N116 | R:R:W106 | 4.52 | Yes | Yes | 1 | 5 | 8 |
| 141 | R:R:F108 | R:R:V107 | 7.87 | Yes | No | 0 | 7 | 2 |
| 142 | R:R:F108 | R:R:M112 | 4.98 | Yes | No | 1 | 7 | 6 |
| 143 | R:R:E110 | R:R:K114 | 12.15 | No | No | 0 | 3 | 7 |
| 144 | R:R:E110 | R:R:M179 | 10.83 | No | Yes | 0 | 3 | 4 |
| 145 | R:R:C113 | R:R:M179 | 4.86 | No | Yes | 1 | 9 | 4 |
| 146 | R:R:C113 | R:R:C198 | 7.28 | No | No | 1 | 9 | 9 |
| 147 | R:R:L115 | R:R:V119 | 4.47 | No | No | 0 | 5 | 5 |
| 148 | R:R:N116 | R:R:P117 | 3.26 | Yes | No | 0 | 5 | 6 |
| 149 | R:R:N116 | R:R:Q120 | 13.2 | Yes | Yes | 1 | 5 | 6 |
| 150 | R:R:F173 | R:R:P117 | 5.78 | Yes | No | 0 | 5 | 6 |
| 151 | R:R:P117 | R:R:Q177 | 14.21 | No | No | 0 | 6 | 5 |
| 152 | R:R:F118 | R:R:V122 | 7.87 | No | No | 5 | 6 | 5 |
| 153 | R:R:F118 | R:R:V167 | 3.93 | No | No | 5 | 6 | 4 |
| 154 | R:R:F118 | R:R:L174 | 19.49 | No | No | 0 | 6 | 5 |
| 155 | R:R:C121 | R:R:S170 | 3.44 | No | No | 0 | 4 | 7 |
| 156 | R:R:C121 | R:R:F173 | 9.78 | No | Yes | 0 | 4 | 5 |
| 157 | R:R:V122 | R:R:V167 | 4.81 | No | No | 5 | 5 | 4 |
| 158 | R:R:S170 | R:R:V122 | 4.85 | No | No | 0 | 7 | 5 |
| 159 | R:R:I124 | R:R:W276 | 7.05 | Yes | Yes | 1 | 7 | 8 |
| 160 | R:R:A166 | R:R:T125 | 3.36 | No | No | 0 | 8 | 6 |
| 161 | R:R:V126 | R:R:W163 | 22.07 | No | No | 0 | 7 | 9 |
| 162 | R:R:I128 | R:R:P223 | 3.39 | No | No | 0 | 7 | 9 |
| 163 | R:R:I128 | R:R:W276 | 5.87 | No | Yes | 0 | 7 | 8 |
| 164 | R:R:F129 | R:R:I162 | 7.54 | No | No | 0 | 7 | 7 |
| 165 | R:R:F129 | R:R:L165 | 7.31 | No | No | 0 | 7 | 5 |
| 166 | R:R:L131 | R:R:N316 | 4.12 | No | No | 2 | 8 | 9 |
| 167 | R:R:L131 | R:R:Y320 | 3.52 | No | Yes | 2 | 8 | 9 |
| 168 | R:R:P223 | R:R:V132 | 3.53 | No | No | 0 | 9 | 7 |
| 169 | R:R:I134 | R:R:R138 | 6.26 | No | No | 2 | 8 | 9 |
| 170 | R:R:I134 | R:R:Y320 | 3.63 | No | Yes | 2 | 8 | 9 |
| 171 | R:R:A135 | R:R:I227 | 4.87 | No | No | 2 | 9 | 8 |
| 172 | R:R:A135 | R:R:Y231 | 5.34 | No | Yes | 2 | 9 | 9 |
| 173 | R:R:C230 | R:R:V136 | 3.42 | Yes | No | 0 | 7 | 6 |
| 174 | R:R:R138 | R:R:Y231 | 7.2 | No | Yes | 2 | 9 | 9 |
| 175 | R:R:R138 | R:R:Y320 | 5.14 | No | Yes | 2 | 9 | 9 |
| 176 | R:R:H139 | R:R:Q140 | 3.71 | Yes | No | 0 | 7 | 4 |
| 177 | R:R:H139 | R:R:I143 | 7.95 | Yes | Yes | 6 | 7 | 6 |
| 178 | R:R:C230 | R:R:H139 | 4.42 | Yes | Yes | 0 | 7 | 7 |
| 179 | R:R:H139 | R:R:K233 | 7.86 | Yes | No | 6 | 7 | 4 |
| 180 | R:R:N144 | R:R:Q140 | 6.6 | No | No | 0 | 6 | 4 |
| 181 | R:R:L141 | R:R:W148 | 6.83 | No | No | 0 | 8 | 5 |
| 182 | R:R:I142 | R:R:I234 | 5.89 | No | No | 0 | 8 | 8 |
| 183 | R:R:I143 | R:R:N144 | 7.08 | Yes | No | 0 | 6 | 6 |
| 184 | R:R:I143 | R:R:K233 | 7.27 | Yes | No | 6 | 6 | 4 |
| 185 | R:R:P145 | R:R:R146 | 8.65 | No | No | 0 | 8 | 6 |
| 186 | R:R:H154 | R:R:N151 | 20.41 | No | No | 0 | 4 | 7 |
| 187 | R:R:P172 | R:R:Y211 | 19.47 | No | No | 0 | 8 | 6 |
| 188 | R:R:F173 | R:R:Y211 | 7.22 | Yes | No | 0 | 5 | 6 |
| 189 | R:R:I175 | R:R:Y176 | 10.88 | No | No | 0 | 4 | 5 |
| 190 | R:R:F202 | R:R:Y176 | 11.35 | Yes | No | 1 | 5 | 5 |
| 191 | R:R:P203 | R:R:Y176 | 8.34 | No | No | 1 | 5 | 5 |
| 192 | R:R:D200 | R:R:Q177 | 3.92 | Yes | No | 0 | 4 | 5 |
| 193 | R:R:T180 | R:R:V178 | 3.17 | No | No | 0 | 3 | 5 |
| 194 | R:R:Q201 | R:R:V178 | 4.3 | No | No | 0 | 3 | 5 |
| 195 | R:R:M179 | R:R:Y196 | 5.99 | Yes | No | 0 | 4 | 5 |
| 196 | R:R:C198 | R:R:M179 | 6.48 | No | Yes | 1 | 9 | 4 |
| 197 | R:R:D181 | R:R:Q185 | 5.22 | No | No | 0 | 1 | 2 |
| 198 | R:R:D181 | R:R:Y196 | 4.6 | No | No | 0 | 1 | 5 |
| 199 | R:R:P183 | R:R:V197 | 5.3 | Yes | No | 1 | 1 | 4 |
| 200 | R:R:F199 | R:R:P183 | 4.33 | Yes | Yes | 1 | 3 | 1 |
| 201 | R:R:F184 | R:R:K195 | 7.44 | No | No | 0 | 1 | 3 |
| 202 | R:R:F184 | R:R:V197 | 11.8 | No | No | 0 | 1 | 4 |
| 203 | R:R:N186 | R:R:V187 | 5.91 | No | No | 0 | 1 | 2 |
| 204 | R:R:T188 | R:R:V187 | 3.17 | No | No | 3 | 3 | 2 |
| 205 | R:R:L189 | R:R:Y192 | 5.86 | No | No | 0 | 5 | 1 |
| 206 | R:R:D194 | R:R:K193 | 4.15 | No | No | 0 | 5 | 2 |
| 207 | R:R:F199 | R:R:V197 | 3.93 | Yes | No | 1 | 3 | 4 |
| 208 | R:R:F199 | R:R:Q201 | 3.51 | Yes | No | 0 | 3 | 3 |
| 209 | R:R:D200 | R:R:F202 | 8.36 | Yes | Yes | 1 | 4 | 5 |
| 210 | R:R:D200 | R:R:R208 | 13.1 | Yes | Yes | 1 | 4 | 4 |
| 211 | R:R:F202 | R:R:P203 | 4.33 | Yes | No | 1 | 5 | 5 |
| 212 | R:R:F202 | R:R:R208 | 19.24 | Yes | Yes | 1 | 5 | 4 |
| 213 | R:R:S204 | R:R:S206 | 3.26 | No | No | 0 | 3 | 1 |
| 214 | R:R:H207 | R:R:S204 | 11.16 | No | No | 0 | 3 | 3 |
| 215 | R:R:D205 | R:R:N289 | 8.08 | No | No | 0 | 1 | 4 |
| 216 | R:R:D287 | R:R:R208 | 4.76 | No | Yes | 1 | 4 | 4 |
| 217 | R:R:L209 | R:R:W288 | 7.97 | No | No | 0 | 3 | 4 |
| 218 | R:R:D287 | R:R:T212 | 13.01 | No | No | 1 | 4 | 5 |
| 219 | R:R:T213 | R:R:W288 | 3.64 | No | No | 0 | 5 | 4 |
| 220 | R:R:L215 | R:R:L218 | 4.15 | No | No | 0 | 5 | 5 |
| 221 | R:R:L215 | R:R:Q219 | 6.65 | No | No | 1 | 5 | 6 |
| 222 | R:R:L216 | R:R:Y220 | 3.52 | No | Yes | 7 | 6 | 8 |
| 223 | R:R:F221 | R:R:L216 | 7.31 | No | No | 7 | 5 | 6 |
| 224 | R:R:L216 | R:R:T284 | 5.9 | No | No | 0 | 6 | 6 |
| 225 | R:R:F221 | R:R:Y220 | 6.19 | No | Yes | 7 | 5 | 8 |
| 226 | R:R:L224 | R:R:Y220 | 4.69 | No | Yes | 0 | 6 | 8 |
| 227 | R:R:L277 | R:R:Y220 | 5.86 | No | Yes | 0 | 6 | 8 |
| 228 | R:R:T280 | R:R:Y220 | 18.73 | No | Yes | 0 | 8 | 8 |
| 229 | R:R:G222 | R:R:P223 | 4.06 | No | No | 0 | 5 | 9 |
| 230 | R:R:F272 | R:R:L224 | 4.87 | No | No | 0 | 9 | 6 |
| 231 | R:R:I227 | R:R:Y231 | 10.88 | No | Yes | 2 | 8 | 9 |
| 232 | R:R:L265 | R:R:Y231 | 11.72 | No | Yes | 0 | 8 | 9 |
| 233 | R:R:V269 | R:R:Y231 | 5.05 | No | Yes | 0 | 8 | 9 |
| 234 | R:R:F232 | R:R:I236 | 10.05 | No | No | 0 | 4 | 3 |
| 235 | R:R:K239 | R:R:Y235 | 4.78 | No | No | 0 | 4 | 5 |
| 236 | R:R:N262 | R:R:Y235 | 8.14 | No | No | 0 | 6 | 5 |
| 237 | R:R:R237 | R:R:R240 | 5.33 | No | No | 0 | 5 | 4 |
| 238 | R:R:R237 | R:R:R241 | 3.2 | No | No | 0 | 5 | 6 |
| 239 | R:R:L238 | R:R:R241 | 3.64 | No | No | 0 | 8 | 6 |
| 240 | R:R:R241 | R:R:R254 | 13.86 | No | Yes | 0 | 6 | 4 |
| 241 | R:R:N242 | R:R:R254 | 13.26 | No | Yes | 0 | 3 | 4 |
| 242 | R:R:M245 | R:R:M248 | 5.78 | No | No | 0 | 5 | 4 |
| 243 | R:R:M245 | R:R:R254 | 14.89 | No | Yes | 0 | 5 | 4 |
| 244 | R:R:M248 | R:R:N251 | 4.21 | No | No | 0 | 4 | 3 |
| 245 | R:R:K252 | R:R:R249 | 3.71 | Yes | No | 0 | 2 | 4 |
| 246 | R:R:K252 | R:R:Y253 | 9.55 | Yes | No | 0 | 2 | 3 |
| 247 | R:R:E257 | R:R:R254 | 9.3 | No | Yes | 0 | 6 | 4 |
| 248 | R:R:E257 | R:R:T258 | 4.23 | No | No | 0 | 6 | 7 |
| 249 | R:R:N262 | R:R:T258 | 4.39 | No | No | 0 | 6 | 7 |
| 250 | R:R:L323 | R:R:M264 | 9.9 | No | No | 0 | 6 | 8 |
| 251 | R:R:F319 | R:R:S267 | 11.89 | Yes | No | 0 | 8 | 5 |
| 252 | R:R:F319 | R:R:I268 | 7.54 | Yes | No | 0 | 8 | 7 |
| 253 | R:R:I268 | R:R:Y320 | 3.63 | No | Yes | 0 | 7 | 9 |
| 254 | R:R:A271 | R:R:V315 | 3.39 | No | No | 0 | 7 | 6 |
| 255 | R:R:F272 | R:R:W276 | 11.02 | No | Yes | 0 | 9 | 8 |
| 256 | R:R:C275 | R:R:W276 | 7.84 | No | Yes | 1 | 8 | 8 |
| 257 | R:R:C275 | R:R:S312 | 6.89 | No | No | 1 | 8 | 9 |
| 258 | R:R:S312 | R:R:W276 | 7.41 | No | Yes | 1 | 9 | 8 |
| 259 | R:R:L277 | R:R:P278 | 4.93 | No | No | 0 | 6 | 9 |
| 260 | R:R:C305 | R:R:P278 | 3.77 | No | No | 0 | 6 | 9 |
| 261 | R:R:P278 | R:R:T308 | 3.5 | No | No | 0 | 9 | 7 |
| 262 | R:R:L279 | R:R:N283 | 8.24 | No | Yes | 0 | 7 | 5 |
| 263 | R:R:F282 | R:R:H298 | 5.66 | Yes | No | 0 | 5 | 3 |
| 264 | R:R:F282 | R:R:F302 | 12.86 | Yes | No | 1 | 5 | 5 |
| 265 | R:R:T284 | R:R:W288 | 4.85 | No | No | 0 | 6 | 4 |
| 266 | R:R:H290 | R:R:Q291 | 3.71 | No | No | 0 | 4 | 2 |
| 267 | R:R:L307 | R:R:M310 | 8.48 | No | Yes | 1 | 5 | 7 |
| 268 | R:R:I311 | R:R:M310 | 4.37 | No | Yes | 0 | 6 | 7 |
| 269 | R:R:F319 | R:R:I318 | 3.77 | Yes | No | 0 | 8 | 6 |
| 270 | R:R:F319 | R:R:L323 | 6.09 | Yes | No | 0 | 8 | 6 |
| 271 | R:R:N324 | R:R:N326 | 12.26 | No | No | 0 | 8 | 6 |
| 272 | R:R:F327 | R:R:N324 | 12.08 | Yes | No | 0 | 8 | 8 |
| 273 | R:R:K325 | R:R:N326 | 6.99 | No | No | 0 | 5 | 6 |
| 274 | R:R:L82 | R:R:S85 | 3 | Yes | No | 0 | 9 | 8 |
| 275 | R:R:L266 | R:R:V270 | 2.98 | No | No | 0 | 5 | 5 |
| 276 | R:R:N152 | R:R:V74 | 2.96 | No | No | 0 | 2 | 6 |
| 277 | R:R:L102 | R:R:T101 | 2.95 | No | No | 0 | 4 | 4 |
| 278 | R:R:I65 | R:R:I79 | 2.94 | No | No | 0 | 7 | 8 |
| 279 | R:R:I143 | R:R:I234 | 2.94 | Yes | No | 0 | 6 | 8 |
| 280 | R:R:A294 | R:R:H298 | 2.93 | No | No | 0 | 1 | 3 |
| 281 | R:R:I64 | R:R:K67 | 2.91 | No | No | 0 | 7 | 5 |
| 282 | R:R:P150 | R:R:R149 | 2.88 | No | No | 0 | 6 | 5 |
| 283 | R:R:I64 | R:R:L331 | 2.85 | No | Yes | 0 | 7 | 5 |
| 284 | R:R:I159 | R:R:L78 | 2.85 | No | Yes | 0 | 7 | 7 |
| 285 | R:R:I162 | R:R:L133 | 2.85 | No | No | 0 | 7 | 7 |
| 286 | R:R:I281 | R:R:L277 | 2.85 | No | No | 0 | 5 | 6 |
| 287 | R:R:L300 | R:R:M39 | 2.83 | No | No | 0 | 2 | 4 |
| 288 | R:R:I261 | R:R:N262 | 2.83 | No | No | 0 | 7 | 6 |
| 289 | R:R:M71 | R:R:N152 | 2.8 | No | No | 0 | 7 | 2 |
| 290 | L:L:H26 | L:L:S22 | 2.79 | No | No | 0 | 0 | 0 |
| 291 | R:R:L303 | R:R:L43 | 2.77 | No | No | 0 | 4 | 7 |
| 292 | R:R:A44 | R:R:F98 | 2.77 | No | No | 0 | 5 | 6 |
| 293 | R:R:L214 | R:R:L218 | 2.77 | No | No | 0 | 4 | 5 |
| 294 | R:R:L300 | R:R:L304 | 2.77 | No | No | 0 | 2 | 6 |
| 295 | R:R:L87 | R:R:N58 | 2.75 | No | Yes | 0 | 8 | 9 |
| 296 | R:R:L300 | R:R:N297 | 2.75 | No | No | 0 | 2 | 3 |
| 297 | R:R:F84 | R:R:V80 | 2.62 | No | No | 0 | 6 | 5 |
| 298 | R:R:F226 | R:R:V132 | 2.62 | No | No | 0 | 6 | 7 |
| 299 | R:R:F319 | R:R:V315 | 2.62 | Yes | No | 0 | 8 | 6 |
| 300 | R:R:F129 | R:R:T125 | 2.59 | No | No | 0 | 7 | 6 |
| 301 | R:R:I143 | R:R:R237 | 2.51 | Yes | No | 0 | 6 | 5 |
| 302 | R:R:F322 | R:R:I318 | 2.51 | No | No | 0 | 6 | 6 |
| 303 | R:R:F282 | R:R:L301 | 2.44 | Yes | No | 0 | 5 | 6 |
| 304 | R:R:F322 | R:R:L331 | 2.44 | No | Yes | 0 | 6 | 5 |
| 305 | R:R:F28 | R:R:N30 | 2.42 | Yes | No | 0 | 6 | 6 |
| 306 | R:R:Q328 | R:R:R260 | 2.34 | No | No | 0 | 8 | 7 |
| 307 | R:R:F232 | R:R:Y235 | 2.06 | No | No | 0 | 4 | 5 |
| 308 | R:R:C314 | R:R:G54 | 1.96 | No | No | 0 | 8 | 7 |
| 309 | R:R:A60 | R:R:G57 | 1.95 | No | No | 0 | 6 | 8 |
| 310 | L:L:A23 | L:L:P5 | 1.87 | No | No | 0 | 0 | 0 |
| 311 | L:L:A12 | L:L:P13 | 1.87 | No | No | 0 | 0 | 0 |
| 312 | R:R:G158 | R:R:V157 | 1.84 | No | No | 0 | 5 | 3 |
| 313 | R:R:G158 | R:R:V161 | 1.84 | No | No | 0 | 5 | 4 |
| 314 | R:R:A135 | R:R:C230 | 1.81 | No | Yes | 0 | 9 | 7 |
| 315 | R:R:G48 | R:R:I51 | 1.76 | No | No | 0 | 5 | 7 |
| 316 | R:R:G109 | R:R:M112 | 1.75 | No | No | 0 | 8 | 6 |
| 317 | R:R:C314 | R:R:V50 | 1.71 | No | No | 0 | 8 | 7 |
| 318 | R:R:A90 | R:R:V55 | 1.7 | No | No | 0 | 7 | 7 |
| 319 | R:R:L171 | R:R:P172 | 1.64 | No | No | 0 | 4 | 8 |
| 320 | R:R:C230 | R:R:I229 | 1.64 | Yes | No | 0 | 7 | 4 |
| 321 | R:R:N144 | R:R:P145 | 1.63 | No | No | 0 | 6 | 8 |
| 322 | R:R:S169 | R:R:S170 | 1.63 | No | No | 0 | 5 | 7 |
| 323 | R:R:T213 | R:R:V217 | 1.59 | No | No | 0 | 5 | 5 |
| 324 | R:R:A60 | R:R:L331 | 1.58 | No | Yes | 0 | 6 | 5 |
| 325 | R:R:A90 | R:R:L94 | 1.58 | No | Yes | 0 | 7 | 7 |
| 326 | R:R:A309 | R:R:L279 | 1.58 | No | No | 0 | 8 | 7 |
| 327 | R:R:E137 | R:R:P150 | 1.57 | Yes | No | 0 | 8 | 6 |
| 328 | R:R:E182 | R:R:P183 | 1.57 | No | Yes | 0 | 1 | 1 |
| 329 | R:R:K252 | R:R:S255 | 1.53 | Yes | No | 0 | 2 | 4 |
| 330 | L:L:A14 | L:L:E15 | 1.51 | No | No | 0 | 0 | 0 |
| 331 | R:R:M39 | R:R:T42 | 1.51 | No | No | 0 | 4 | 5 |
| 332 | R:R:F327 | R:R:G321 | 1.51 | Yes | No | 0 | 8 | 7 |
| 333 | R:R:L88 | R:R:S123 | 1.5 | No | No | 0 | 7 | 8 |
| 334 | R:R:L66 | R:R:V80 | 1.49 | No | No | 0 | 4 | 5 |
| 335 | R:R:I159 | R:R:I77 | 1.47 | No | No | 0 | 7 | 5 |
| 336 | R:R:G147 | R:R:W148 | 1.41 | No | No | 0 | 4 | 5 |
| 337 | R:R:K193 | R:R:L189 | 1.41 | No | No | 0 | 2 | 5 |
| 338 | R:R:M244 | R:R:N243 | 1.4 | No | No | 0 | 2 | 4 |
| 339 | R:R:D250 | R:R:K252 | 1.38 | No | Yes | 0 | 3 | 2 |
| 340 | R:R:N242 | R:R:N243 | 1.36 | No | No | 0 | 3 | 4 |
| 341 | R:R:A191 | R:R:Y192 | 1.33 | No | No | 0 | 3 | 1 |
| 342 | R:R:F108 | R:R:V99 | 1.31 | Yes | No | 0 | 7 | 4 |
| 343 | R:R:S210 | R:R:Y211 | 1.27 | No | No | 0 | 4 | 6 |
| 344 | R:R:S256 | R:R:Y253 | 1.27 | No | No | 0 | 4 | 3 |
| 345 | R:R:F228 | R:R:I229 | 1.26 | No | No | 0 | 5 | 4 |
| 346 | R:R:F334 | R:R:L331 | 1.22 | No | Yes | 0 | 5 | 5 |
| 347 | R:R:N151 | R:R:R153 | 1.21 | No | No | 0 | 7 | 5 |
| 348 | R:R:N251 | R:R:R249 | 1.21 | No | No | 0 | 3 | 4 |
| 349 | R:R:D330 | R:R:R329 | 1.19 | No | No | 0 | 5 | 5 |
| 350 | L:L:Y1 | R:R:D205 | 1.15 | Yes | No | 0 | 0 | 1 |
| 351 | R:R:F333 | R:R:F334 | 1.07 | No | No | 0 | 4 | 5 |
| 352 | R:R:R153 | R:R:Y156 | 1.03 | No | No | 0 | 5 | 3 |
| Color | ConSurf Grade |
| No Conservation data available | |
| 1 | |
| 2 | |
| 3 | |
| 4 | |
| 5 | |
| 6 | |
| 7 | |
| 8 | |
| 9 |
Index: hub id, click on each number to highlight the corresponding hub in the 3D visualization.
Hub: the hub being considered.
Avg Int. Strength: the average interaction strength of all the links of the corresponding hub.
Num Of Links: the number of links of the corresponding hub.
Community: the id of the community the link belong to, otherwise 0.
ConSurf: this column reports the ConSurf conservation grades of each hub.
| Index | Hub | Avg Int. Strength | Num Of Links | Community | ConSurf |
|---|---|---|---|---|---|
| 1 | L:L:Y1 | 6.85167 | 6 | 1 | 0 |
| 2 | L:L:P2 | 6.32 | 4 | 1 | 0 |
| 3 | L:L:Y20 | 6.07 | 4 | 3 | 0 |
| 4 | L:L:Y21 | 9.162 | 5 | 3 | 0 |
| 5 | L:L:R33 | 10.0225 | 4 | 1 | 0 |
| 6 | L:L:Q34 | 7.42 | 5 | 1 | 0 |
| 7 | L:L:R35 | 11.27 | 5 | 1 | 0 |
| 8 | L:L:?36 | 9.01125 | 8 | 1 | 0 |
| 9 | R:R:F28 | 5.65 | 4 | 3 | 6 |
| 10 | R:R:E29 | 10.0375 | 4 | 0 | 4 |
| 11 | R:R:Y47 | 6.4 | 6 | 1 | 7 |
| 12 | R:R:N58 | 9.268 | 5 | 4 | 9 |
| 13 | R:R:L78 | 4.835 | 4 | 0 | 7 |
| 14 | R:R:L82 | 6.934 | 5 | 2 | 9 |
| 15 | R:R:V89 | 4.46 | 4 | 1 | 7 |
| 16 | R:R:M92 | 6.1275 | 4 | 1 | 5 |
| 17 | R:R:L94 | 5.58 | 6 | 1 | 7 |
| 18 | R:R:F96 | 9.405 | 4 | 1 | 6 |
| 19 | R:R:T97 | 6.47 | 4 | 1 | 6 |
| 20 | R:R:W106 | 9.96 | 5 | 1 | 8 |
| 21 | R:R:F108 | 8.584 | 5 | 1 | 7 |
| 22 | R:R:N116 | 8.17667 | 6 | 1 | 5 |
| 23 | R:R:Q120 | 7.05167 | 6 | 1 | 6 |
| 24 | R:R:I124 | 5.24 | 4 | 1 | 7 |
| 25 | R:R:E137 | 5.24 | 4 | 0 | 8 |
| 26 | R:R:H139 | 5.985 | 4 | 6 | 7 |
| 27 | R:R:I143 | 5.55 | 5 | 6 | 6 |
| 28 | R:R:F173 | 7.03 | 4 | 0 | 5 |
| 29 | R:R:M179 | 7.04 | 4 | 1 | 4 |
| 30 | R:R:P183 | 3.8425 | 4 | 1 | 1 |
| 31 | R:R:F199 | 4.596 | 5 | 1 | 3 |
| 32 | R:R:D200 | 8.3575 | 4 | 1 | 4 |
| 33 | R:R:F202 | 10.82 | 4 | 1 | 5 |
| 34 | R:R:R208 | 12.3625 | 4 | 1 | 4 |
| 35 | R:R:Y220 | 7.798 | 5 | 7 | 8 |
| 36 | R:R:C230 | 2.8225 | 4 | 0 | 7 |
| 37 | R:R:Y231 | 8.038 | 5 | 2 | 9 |
| 38 | R:R:K252 | 4.0425 | 4 | 0 | 2 |
| 39 | R:R:R254 | 12.8275 | 4 | 0 | 4 |
| 40 | R:R:W276 | 7.838 | 5 | 1 | 8 |
| 41 | R:R:F282 | 7.11 | 4 | 1 | 5 |
| 42 | R:R:N283 | 7.1475 | 4 | 1 | 5 |
| 43 | R:R:H306 | 5.3275 | 4 | 1 | 6 |
| 44 | R:R:M310 | 7.898 | 5 | 1 | 7 |
| 45 | R:R:F319 | 6.382 | 5 | 0 | 8 |
| 46 | R:R:Y320 | 3.845 | 6 | 2 | 9 |
| 47 | R:R:F327 | 6.175 | 4 | 0 | 8 |
| 48 | R:R:L331 | 2.0225 | 4 | 0 | 5 |
| Color | ConSurf Grade |
| No Conservation data available | |
| 1 | |
| 2 | |
| 3 | |
| 4 | |
| 5 | |
| 6 | |
| 7 | |
| 8 | |
| 9 |
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
Recurrence: the relative Recurrence in the pool of shortest paths.
Int. Strength: the interaction strength between the two nodes.
Hub1?, Hub2?: "Yes" if the corresponding node has more than 3 links, otherwise "No".
Community: the id of the community the link belong to, otherwise 0.
ConSurf1, ConSurf2: these columns report the ConSurf conservation grades of the two nodes involved in a link.
| Index | Node1 | Node2 | Recurrence | Int. Strength | Hub1? | Hub2? | Community | ConSurf1 | ConSurf2 |
|---|---|---|---|---|---|---|---|---|---|
| 1 | L:L:I31 | L:L:Y1 | 14.4046 | 3.63 | No | Yes | 1 | 0 | 0 |
| 2 | L:L:Y1 | R:R:F199 | 26.1762 | 6.19 | Yes | Yes | 1 | 0 | 3 |
| 3 | L:L:Y1 | R:R:R208 | 37.2814 | 12.35 | Yes | Yes | 1 | 0 | 4 |
| 4 | L:L:K4 | L:L:Y27 | 13.4172 | 8.36 | No | No | 0 | 0 | 0 |
| 5 | L:L:I31 | L:L:Y27 | 13.8374 | 12.09 | No | No | 0 | 0 | 0 |
| 6 | L:L:K4 | R:R:N186 | 11.2997 | 8.39 | No | No | 0 | 0 | 1 |
| 7 | R:R:N186 | R:R:V187 | 10.5918 | 5.91 | No | No | 0 | 1 | 2 |
| 8 | L:L:L24 | R:R:E29 | 18.7763 | 9.28 | No | Yes | 0 | 0 | 4 |
| 9 | L:L:I28 | L:L:L24 | 19.5442 | 5.71 | No | No | 0 | 0 | 0 |
| 10 | L:L:I28 | R:R:F184 | 22.5953 | 8.79 | No | No | 0 | 0 | 1 |
| 11 | R:R:F184 | R:R:V197 | 21.5748 | 11.8 | No | No | 0 | 1 | 4 |
| 12 | R:R:F199 | R:R:V197 | 21.9288 | 3.93 | Yes | No | 1 | 3 | 4 |
| 13 | L:L:Q34 | R:R:Q120 | 11.2831 | 6.4 | Yes | Yes | 1 | 0 | 6 |
| 14 | R:R:N116 | R:R:W106 | 10.8919 | 4.52 | Yes | Yes | 1 | 5 | 8 |
| 15 | R:R:D287 | R:R:R208 | 40.4069 | 4.76 | No | Yes | 1 | 4 | 4 |
| 16 | L:L:R35 | R:R:D287 | 40.9389 | 8.34 | Yes | No | 1 | 0 | 4 |
| 17 | L:L:?36 | R:R:I124 | 69.6653 | 6.03 | Yes | Yes | 1 | 0 | 7 |
| 18 | L:L:?36 | R:R:H306 | 20.5647 | 7.61 | Yes | Yes | 1 | 0 | 6 |
| 19 | R:R:H306 | R:R:L94 | 10.1819 | 3.86 | Yes | Yes | 1 | 6 | 7 |
| 20 | R:R:H306 | R:R:Y47 | 11.8793 | 4.36 | Yes | Yes | 1 | 6 | 7 |
| 21 | R:R:I124 | R:R:V89 | 70.81 | 4.61 | Yes | Yes | 1 | 7 | 7 |
| 22 | R:R:T313 | R:R:V89 | 99.9069 | 3.17 | No | Yes | 0 | 8 | 7 |
| 23 | R:R:D86 | R:R:T313 | 99.9338 | 5.78 | No | No | 0 | 9 | 8 |
| 24 | R:R:D86 | R:R:N316 | 100 | 4.04 | No | No | 0 | 9 | 9 |
| 25 | R:R:L82 | R:R:N316 | 50.6862 | 15.1 | Yes | No | 2 | 9 | 9 |
| 26 | R:R:L82 | R:R:Y320 | 49.7175 | 3.52 | Yes | Yes | 2 | 9 | 9 |
| 27 | R:R:I268 | R:R:Y320 | 18.9501 | 3.63 | No | Yes | 0 | 7 | 9 |
| 28 | R:R:F319 | R:R:I268 | 17.8738 | 7.54 | Yes | No | 0 | 8 | 7 |
| 29 | R:R:F319 | R:R:I318 | 11.2769 | 3.77 | Yes | No | 0 | 8 | 6 |
| 30 | R:R:F322 | R:R:I318 | 10.1633 | 2.51 | No | No | 0 | 6 | 6 |
| 31 | R:R:L131 | R:R:N316 | 49.3014 | 4.12 | No | No | 2 | 8 | 9 |
| 32 | R:R:L131 | R:R:Y320 | 49.0509 | 3.52 | No | Yes | 2 | 8 | 9 |
| 33 | R:R:I79 | R:R:Y320 | 17.9152 | 3.63 | No | Yes | 0 | 8 | 9 |
| 34 | R:R:I65 | R:R:I79 | 16.8264 | 2.94 | No | No | 0 | 7 | 8 |
| 35 | R:R:F327 | R:R:I65 | 12.4278 | 6.28 | Yes | No | 0 | 8 | 7 |
| 36 | R:R:I134 | R:R:Y320 | 25.075 | 3.63 | No | Yes | 2 | 8 | 9 |
| 37 | R:R:I134 | R:R:L78 | 26.4764 | 5.71 | No | Yes | 0 | 8 | 7 |
| 38 | R:R:I159 | R:R:L78 | 10.2068 | 2.85 | No | Yes | 0 | 7 | 7 |
| 39 | R:R:C93 | R:R:Q120 | 32.3301 | 7.63 | No | Yes | 1 | 7 | 6 |
| 40 | R:R:F108 | R:R:W106 | 10.0578 | 18.04 | Yes | Yes | 1 | 7 | 8 |
| 41 | R:R:M103 | R:R:V107 | 10.2006 | 9.13 | No | No | 0 | 3 | 2 |
| 42 | R:R:F108 | R:R:V107 | 12.8873 | 7.87 | Yes | No | 0 | 7 | 2 |
| 43 | R:R:C113 | R:R:W106 | 12.7652 | 10.45 | No | Yes | 1 | 9 | 8 |
| 44 | R:R:I124 | R:R:W276 | 24.4892 | 7.05 | Yes | Yes | 1 | 7 | 8 |
| 45 | R:R:R138 | R:R:Y320 | 47.0245 | 5.14 | No | Yes | 2 | 9 | 9 |
| 46 | R:R:R138 | R:R:Y231 | 48.5314 | 7.2 | No | Yes | 2 | 9 | 9 |
| 47 | R:R:A135 | R:R:Y231 | 44.5985 | 5.34 | No | Yes | 2 | 9 | 9 |
| 48 | R:R:A135 | R:R:C230 | 43.7043 | 1.81 | No | Yes | 0 | 9 | 7 |
| 49 | R:R:C230 | R:R:H139 | 39.7384 | 4.42 | Yes | Yes | 0 | 7 | 7 |
| 50 | R:R:H139 | R:R:I143 | 33.4023 | 7.95 | Yes | Yes | 6 | 7 | 6 |
| 51 | R:R:F272 | R:R:W276 | 16.5904 | 11.02 | No | Yes | 0 | 9 | 8 |
| 52 | R:R:F272 | R:R:L224 | 15.5844 | 4.87 | No | No | 0 | 9 | 6 |
| 53 | R:R:L224 | R:R:Y220 | 14.5743 | 4.69 | No | Yes | 0 | 6 | 8 |
| 54 | R:R:I143 | R:R:R237 | 27.3333 | 2.51 | Yes | No | 0 | 6 | 5 |
| 55 | R:R:R237 | R:R:R241 | 25.2696 | 3.2 | No | No | 0 | 5 | 6 |
| 56 | R:R:R241 | R:R:R254 | 23.1459 | 13.86 | No | Yes | 0 | 6 | 4 |
| 57 | R:R:M245 | R:R:R254 | 10.1612 | 14.89 | No | Yes | 0 | 5 | 4 |
| 58 | L:L:?36 | L:L:R35 | 47.5937 | 21.56 | Yes | Yes | 1 | 0 | 0 |
| 59 | R:R:N116 | R:R:Q120 | 18.3209 | 13.2 | Yes | Yes | 1 | 5 | 6 |
| 60 | R:R:C93 | R:R:V89 | 28.9893 | 6.83 | No | Yes | 1 | 7 | 7 |
| 61 | L:L:Q34 | R:R:W106 | 11.8255 | 3.29 | Yes | Yes | 1 | 0 | 8 |
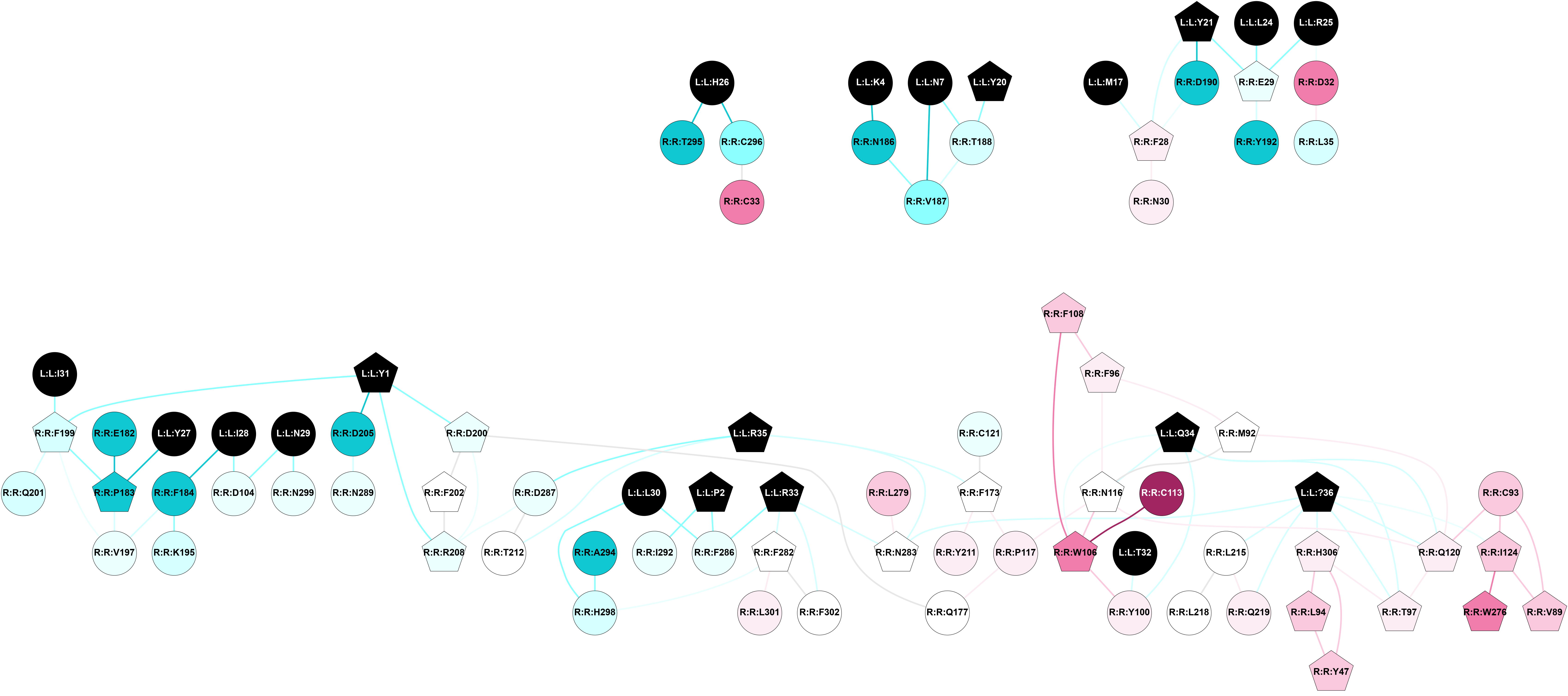
2D representation of the global metapath, ligand(s) interactions and
histograms of path distribution according to several parameters
(click on the image to enlarge it 🔍):
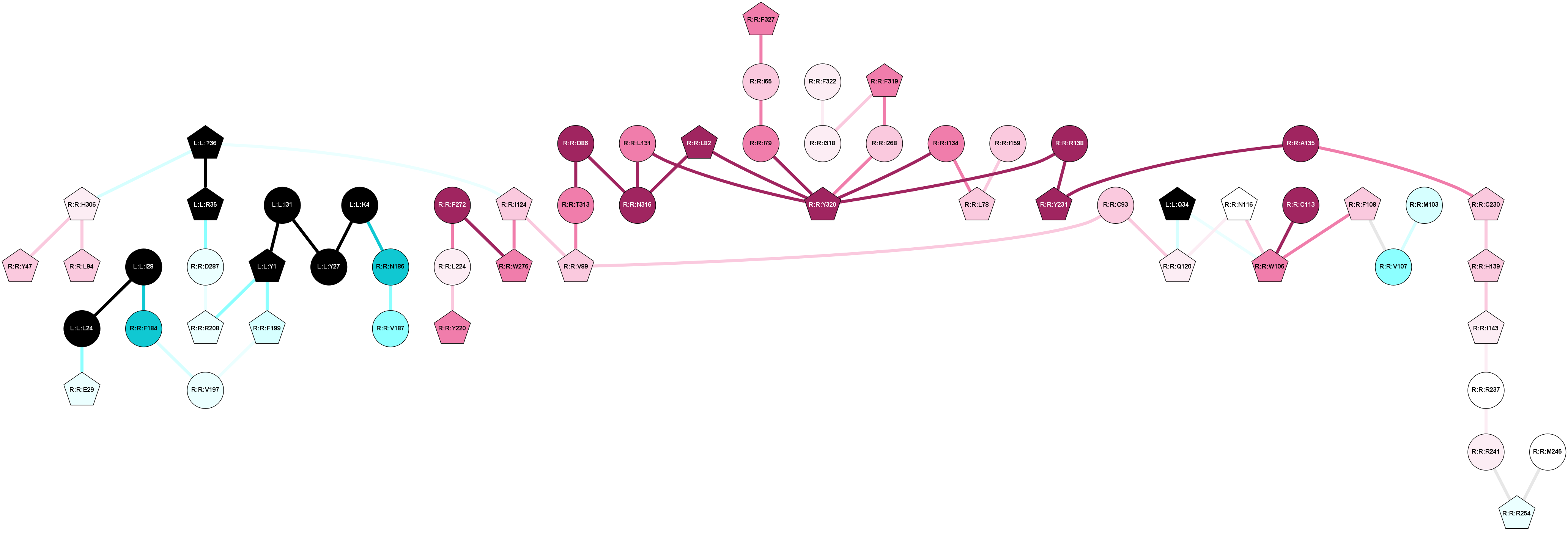
A 2D representation of the global communication in the network.
ConSurf Conservation Grade (See documentation):
n/a 1 2 3 4 5 6 7 8 9
2D representation of the interactions of this orthosteric/allosteric ligand. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Links and nodes colored according to ConSurf Conservation Grade (See documentation): n/a 1 2 3 4 5 6 7 8 9 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Location and physicochemical properties of the interaction partners of this ligand | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Interactions of this ligand | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Similarities between the interactions of this ligand and those of other networks | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
Details about the values in these tables can be found in the corresponding documentation page .
| |||||||||||||||||||||||||||||||||||
| PDBsum | Open PDBsum Page |
| Chain | R |
| Protein | Receptor |
| UniProt | P25929 |
| Sequence | >7X9A_nogp_Chain_R FENDDCHLP LAMIFTLAL AYGAVIILG VSGNLALII IILKQKEMR NVTNILIVN LSFSDLLVA IMCLPFTFV YTLMDHWVF GEAMCKLNP FVQCVSITV SIFSLVLIA VERHQLIIN PRGWRPNNR HAYVGIAVI WVLAVASSL PFLIYQVMT DEPFQNVTL DAYKDKYVC FDQFPSDSH RLSYTTLLL VLQYFGPLC FIFICYFKI YIRLKRRNN MMDKMRDNK YRSSETKRI NIMLLSIVV AFAVCWLPL TIFNTVFDW NHQIIATCN HNLLFLLCH LTAMISTCV NPIFYGFLN KNFQRDLQF FFN Click on each residue to open a popup with some information about it. ConSurf Conservation Grade (See documentation): n/a 1 2 3 4 5 6 7 8 9 |
| This receptor, from the same or other species and bound to the same or other ligands, is also present in the following networks: | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Show | PDB | Class | SubFamily | Type | SubType | Species | Orthosteric Ligand | Other Ligand(s) | Protein Partners | Resolution | Date | DOI |
| 9AUC | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Calcitonin gene-related peptide-1 | - | Gs/β1/γ2; RAMP1 | 2.4 | 2024-04-24 | doi.org/10.1021/acs.biochem.4c00114 | |
| 9AUC (No Gprot) | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Calcitonin gene-related peptide-1 | - | 2.4 | 2024-04-24 | doi.org/10.1021/acs.biochem.4c00114 | ||
| 7TYF | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Amylin | - | Gs/β1/γ2; RAMP1 | 2.2 | 2022-03-23 | doi.org/10.1126/science.abm9609 | |
| 7TYF (No Gprot) | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Amylin | - | 2.2 | 2022-03-23 | doi.org/10.1126/science.abm9609 | ||
| 7TYW | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Calcitonin-1 | - | Gs/β1/γ2; RAMP1 | 3 | 2022-03-23 | doi.org/10.1126/science.abm9609 | |
| 7TYW (No Gprot) | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Calcitonin-1 | - | 3 | 2022-03-23 | doi.org/10.1126/science.abm9609 | ||
| 9BLW | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Cagrilintide backbone (non-acylated) | - | Gs/β1/γ2; RAMP1 | 3.2 | 2025-04-16 | To be published | |
| 9BLW (No Gprot) | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Cagrilintide backbone (non-acylated) | - | 3.2 | 2025-04-16 | To be published | ||
| 9BP3 | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Cagrilintide | - | Gs/β1/γ2; RAMP1 | 2.2 | 2025-04-23 | To be published | |
| 9BP3 (No Gprot) | B1 | Peptide | Calcitonin | CT (AMY1) | Homo sapiens | Cagrilintide | - | 2.2 | 2025-04-23 | To be published | ||
| 7VGX | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | Gi1/β1/γ2 | 3.2 | 2022-02-23 | doi.org/10.1038/s41467-022-28510-6 | |
| 7VGX (No Gprot) | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | 3.2 | 2022-02-23 | doi.org/10.1038/s41467-022-28510-6 | ||
| 5ZBH | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | BMS-193885 | - | - | 3 | 2018-04-25 | doi.org/10.1038/s41586-018-0046-x | |
| 5ZBQ | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | UR-MK-299 | - | - | 2.7 | 2018-04-25 | doi.org/10.1038/s41586-018-0046-x | |
| 7X9A | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | Gi1/β1/γ2 | 3.2 | 2022-05-18 | doi.org/10.1126/sciadv.abm1232 | |
| 7X9A (No Gprot) | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | 3.2 | 2022-05-18 | doi.org/10.1126/sciadv.abm1232 | ||
| 8K6M | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | Gi2/β1/γ1 | 3.3 | 2024-07-31 | doi.org/10.1002/mco2.565 | |
| 8K6M (No Gprot) | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Neuropeptide-Y | - | 3.3 | 2024-07-31 | doi.org/10.1002/mco2.565 | ||
| 8K6O | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Pro-neuropeptide-Y | - | Gi2/β1/γ1 | 3.3 | 2024-07-31 | doi.org/10.1002/mco2.565 | |
| 8K6O (No Gprot) | A | Peptide | Neuropeptide Y | Y1 | Homo sapiens | Pro-neuropeptide-Y | - | 3.3 | 2024-07-31 | doi.org/10.1002/mco2.565 | ||
| 7Y1F | A | Peptide | Opioid | κ | Homo sapiens | Dynorphin | - | Gi1/β1/γ2 | 3.3 | 2023-05-24 | doi.org/10.1093/procel/pwac033 | |
| 7Y1F (No Gprot) | A | Peptide | Opioid | κ | Homo sapiens | Dynorphin | - | 3.3 | 2023-05-24 | doi.org/10.1093/procel/pwac033 | ||
| 7Y12 | A | Orphan | Orphan | MRGPRD | Homo sapiens | β-Alanine | - | Gi1/β1/γ2 | 3.1 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | |
| 7Y12 (No Gprot) | A | Orphan | Orphan | MRGPRD | Homo sapiens | β-Alanine | - | 3.1 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | ||
| 7Y13 | A | Orphan | Orphan | MRGPRD | Homo sapiens | - | - | - | 3.1 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | |
| 7Y14 | A | Orphan | Orphan | MRGPRD | Homo sapiens | β-Alanine | - | - | 3.2 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | |
| 7Y15 | A | Orphan | Orphan | MRGPRD | Homo sapiens | - | - | Gi1/β1/γ2 | 2.9 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | |
| 7Y15 (No Gprot) | A | Orphan | Orphan | MRGPRD | Homo sapiens | - | - | 2.9 | 2022-07-20 | doi.org/10.1038/s42003-022-03668-3 | ||
| 8KGG | A | Nucleotide | P2Y | P2Y10 | Homo sapiens | LPS | - | chim(NtGi2-G13)/β1/γ2 | 3.06 | 2024-07-31 | doi.org/10.1016/j.chembiol.2024.08.005 | |
| 8KGG (No Gprot) | A | Nucleotide | P2Y | P2Y10 | Homo sapiens | LPS | - | 3.06 | 2024-07-31 | doi.org/10.1016/j.chembiol.2024.08.005 | ||
| 6D27 | A | Lipid | Prostanoid | DP2 | Homo sapiens | CAY10471 | - | - | 2.74 | 2018-10-03 | doi.org/10.1016/j.molcel.2018.08.009 | |
You can download a compressed (zip) file with structure(s), 3D outputs (as PyMol and VMD scripts) and numerical data files (as csv and plain text files).
Download 7X9A_nogp.zipYou can click to copy the link of this page to easily come back here later
or use this QR code to link and share this page.
You can also read or download a guide explaining the meaning of all files and numerical data.